El trigo, un cultivo alimentario de importancia mundial, obtiene su rendimiento de factores como el número de espigas por unidad de superficie, el peso de mil granos y el rendimiento de grano por espiga.
por Zhang Nannan, Academia China de Ciencias
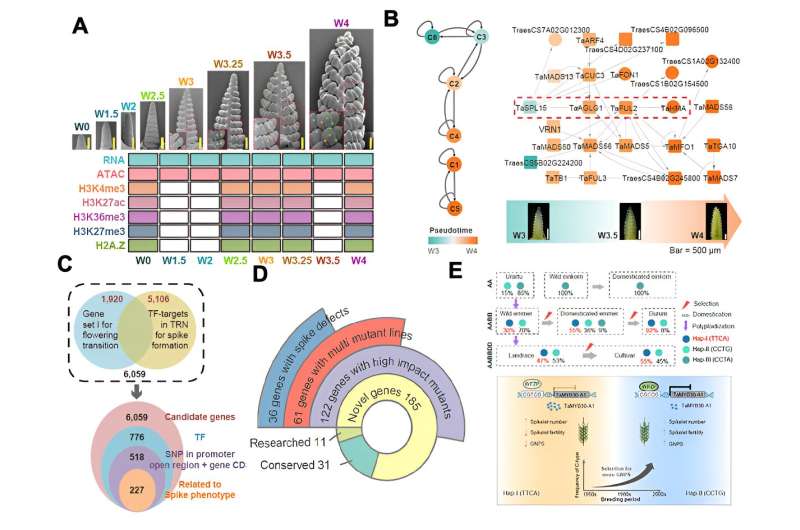
La forma de la espiga afecta directamente el número de espiguillas, el rendimiento de grano y la productividad general del trigo. Descubrir los reguladores clave del desarrollo de la espiga del trigo e investigar sus mecanismos moleculares es muy prometedor para el mejoramiento genético de precisión y la mejora molecular de la morfología de la espiga.
En un estudio publicado en Molecular Plant , investigadores dirigidos por Xiao Jun del Instituto de Genética y Biología del Desarrollo (IGDB) de la Academia China de Ciencias (CAS) presentaron un enfoque innovador. Al integrar ómicas multidimensionales, genética de poblaciones y análisis de funciones genéticas, los investigadores delinearon una estrategia sistemática y eficiente para identificar factores reguladores clave en el desarrollo de la espiga del trigo .
A través de la secuenciación integral de los transcriptomas de la espiga del trigo, la accesibilidad a la cromatina y las modificaciones de las histonas en etapas clave del desarrollo, delinearon el panorama transcripcional y epigenético dinámico de la maduración de la espiga del trigo, que culminó en la construcción de una red reguladora transcripcional (TRN).
Al fusionar las ómicas multidimensionales con la genética de poblaciones, los investigadores identificaron 227 supuestos factores reguladores que influyen en el desarrollo de las espigas, 42 de los cuales ya han sido implicados en la formación de espigas en el trigo o el arroz.
La detección fenotípica de 61 genes nuevos utilizando la biblioteca de mutantes KN9204 reveló 36 mutaciones, incluidas TaMYC2-A1, TaMYB30-A1 y TaWRKY37-A1, que alteran el tiempo de floración o la morfología de las espigas. La caracterización funcional de TaMYB30-A1 subrayó la eficacia de TRN para descifrar la funcionalidad genética y su utilidad práctica en iniciativas de mejoramiento de trigo.
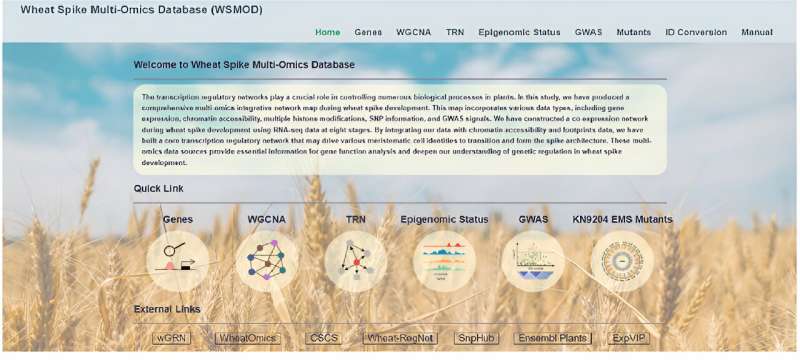
Al mismo tiempo, se lanzó una base de datos multiómica integral para el desarrollo de la espiga del trigo, conocida como WSMOD. Esta plataforma proporciona a los investigadores un cómodo acceso «todo en uno» a diversos servicios, incluida la recuperación de información genética, el análisis de coexpresión, la predicción de TRN, el trazado de mapas epigenéticos y las funciones de búsqueda de bibliotecas de mutantes.
En resumen, este estudio aclara las transformaciones dinámicas que ocurren durante el desarrollo de la espiga del trigo, que están determinadas tanto por la transcripción genética como por la regulación epigenética. Integra datos multidimensionales que incluyen transcriptómica, epigenómica y genética de poblaciones para desentrañar estos procesos. La construcción de una TRN específica para el desarrollo de la espiga de trigo representa una estrategia sistemática, eficiente y precisa para identificar factores regulatorios clave.
Más información: Xuelei Lin et al, Identificación sistémica de reguladores del desarrollo de la espiga del trigo mediante multiómicas integradas, red transcripcional, GWAS y análisis genéticos, Molecular Plant (2024). DOI: 10.1016/j.molp.2024.01.010
Nota editorial:
Este artículo ha sido elaborado con fines divulgativos a partir de información pública y fuentes especializadas, adaptado al enfoque editorial del medio para facilitar su comprensión y contextualización.
