Las rosas, famosas en todo el mundo por su valor ornamental, tienen una larga historia de cría y cultivo, con más de 30.000 cultivares.
por la Universidad Agrícola de NanJing
Las rosas son plantas predominantemente cruzadas y altamente heterocigotas, y los patrones de herencia para la mayoría de los rasgos son difíciles de predecir. Como resultado, el cruzamiento de rosas es extremadamente desafiante.
Los avances recientes en la genética de las rosas, incluido el desarrollo de marcadores moleculares para construir mapas de vínculos genéticos y la secuenciación del genoma , han descubierto factores genéticos clave para rasgos como el olor, la floración continua (CF) y la doble flor (DF). Sin embargo, el locus S que controla la autocompatibilidad (SI) de las rosas no se ha dilucidado completamente, lo cual es esencial para mejorar la reproducción a nivel diploide.
Investigaciones relacionadas han identificado posibles regiones del locus S y enzimas S-RNasa asociadas en el genoma, pero las inconsistencias y los experimentos de polinización limitados dejan sin confirmar la ubicación y función exacta del locus S.
En octubre de 2022, Horticulture Research publicó una perspectiva titulada » La identificación del locus Rosa S proporciona nuevos conocimientos sobre la reproducción y los orígenes silvestres de las rosas de floración continua «.
Este estudio tiene como objetivo identificar el locus S de Rosa que controla la autoincompatibilidad (SI), explorar los vínculos genéticos del locus S con otros rasgos ornamentales, investigar los cambios de frecuencia históricos de alelos S específicos (SC) en cultivares de rosas , e identificar especies silvestres que portan los alelos Sc para inferir orígenes silvestres de antiguos cultivares chinos de CF. Esta investigación potencialmente desentrañará conexiones genéticas clave y ayudará a implementar prácticas de mejoramiento de rosas más precisas y eficientes.
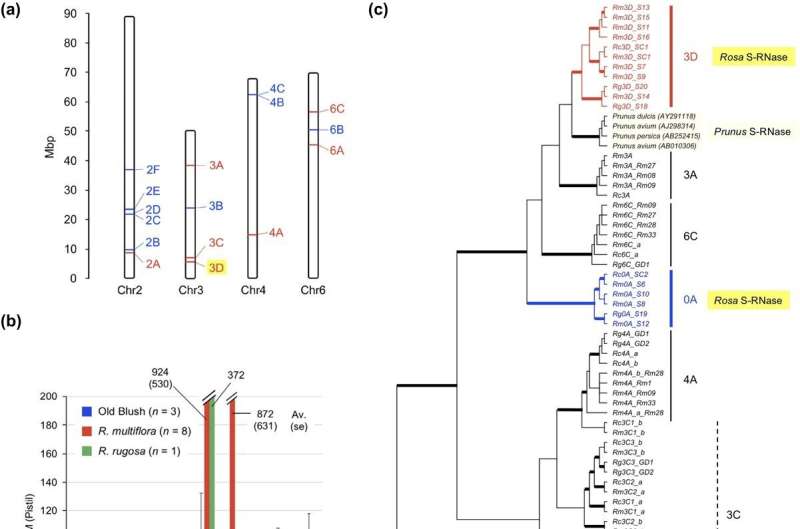
En primer lugar, los investigadores realizaron una búsqueda en todo el genoma de genes S-RNasa candidatos en las bases de datos del genoma de Old Blush, R. multiflora y R. rugosa. Identificaron 16 genes candidatos ubicados en cuatro cromosomas, siete de ellos expresados en el pistilo.
El análisis del árbol filogenético molecular identificó además los genes 3D y 0A como los principales candidatos para la S-RNasa. Y se descubrieron nueve genes adicionales similares a 3D S-RNasa en R. multiflora y R. rugosa. La alta divergencia genética apoyó la hipótesis de que el gen 3D S-RNasa codifica la verdadera S-RNasa que controla el SI en las rosas.
Los investigadores centraron su atención en el gen 0A, que se supone que es alélico de la 3D S-RNasa. El análisis de los transcriptomas de pistilo de siete plantas diploides de R. multiflora sugirió que los genes 0A y 3D son alélicos. El mapeo genético en una población diploide confirmó además que los genes 0A y 3D S-RNasa se asignan a posiciones homólogas en el cromosoma 3.
En segundo lugar, los genes F-box que flanquean la S-RNasa se identificaron mediante la búsqueda en la base de datos del genoma y los transcriptomas de los estambres de Old Blush . Se identificaron 12 genes F-box y una S-RNasa (S C1 ) de la región de 5,3 a 5,8 Mbp. en Chr3 y otros tres genes F-box se identificaron en el contig RC0 que contiene la S C2 S-RNasa.
Amplios experimentos de polinización respaldaron firmemente la hipótesis de que los genes 3D y 0A S-RNasa son el determinante del pistilo del SI en las rosas. Además, el análisis de segregación de una población F 1 indicó un vínculo genético entre el locus S y el gen represor floral KSN. El alelo no funcional ksn es responsable de la característica de la FQ.
Finalmente, el estudio rastreó los alelos S C de antiguos cultivares chinos de CF y se identificaron un total de cinco alelos S (S C1–5 ). La frecuencia de cultivares con S C aumentó dramáticamente después de la introgresión de ksn de los cultivares chinos a los europeos y sigue siendo alta (80%) en los cultivares modernos, lo que sugiere que el genotipado S es útil para un mejoramiento eficaz.
En resumen, este estudio ofrece información valiosa para el mejoramiento de rosas, lo que demuestra la importancia del genotipado S para un mejoramiento efectivo, especialmente en rosas diploides. Proporciona una base para futuros estudios sobre los vínculos genéticos entre el locus S y rasgos como DF, ausencia de espinas y resistencia a enfermedades , y contribuye significativamente a nuestra comprensión de la genética y las prácticas de mejoramiento de las rosas.
Más información: Koji Kawamura et al, La identificación del locus Rosa S proporciona nuevos conocimientos sobre la reproducción y los orígenes silvestres de las rosas de floración continua, Horticulture Research (2022). DOI: 10.1093/hora/uhac155
