Las fresas cultivadas son ahora la base de una industria global multimillonaria.
por Fenómica vegetal
Para enfrentar los desafíos planteados por la creciente demanda, el cambio climático y la escasez de agua y tierra, es necesario recolectar nuevo germoplasma para mejorar los cultivares. Sin embargo, la gestión de colecciones de germoplasma propagado clonalmente, como la fresa, es costosa, mientras que la caracterización genotípica y fenotípica limitada dificulta la utilización eficaz del germoplasma.
Las plataformas de ensamblaje del genoma y genotipado han permitido una comprensión más profunda de la diversidad genética y la genética de los rasgos. El Repositorio Nacional de Germoplasma Clonal (NCGR) del USDA-ARS en Corvallis, Oregon, mantiene la colección de fresas de EE. UU., pero se ha hecho muy poco para explorar la diversidad molecular dentro de la colección nacional de F. ×ananassa de EE. UU.
En mayo de 2022, Horticulture Research publicó un artículo de investigación titulado » Exploración de la diversidad y estructura genética de la colección nacional de fresas cultivadas de EE. UU .».
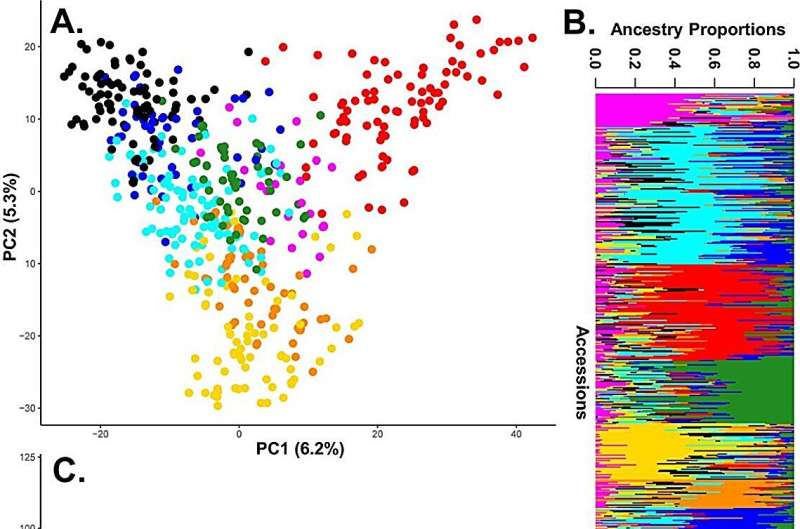
En este estudio, la curación de datos implicó el genotipado de accesiones de fresa utilizando los conjuntos Axiom IStraw35 y Axiom FanaSNP, que identificaron 27,968 y 40,424 marcadores SNP, respectivamente. Los datos de los marcadores compartidos por las dos matrices se analizaron para determinar la calidad de las llamadas, los datos faltantes y las frecuencias de alelos menores, lo que dio como resultado 4033 marcadores para la evaluación estructural, el análisis de la diversidad, la confirmación del pedigrí, el desarrollo de la colección central y la identificación de haplotipos asociados con rasgos deseables.
La estructura de la población se analizó mediante Análisis de Componentes Principales (PCA). La agrupación de K-medias y el algoritmo sNMF identificaron ocho subpoblaciones óptimas. F. ×ananassa tuvo la misma diversidad en todas las regiones geográficas. La agrupación jerárquica utilizando UPGMA identificó de siete a nueve clados principales que también estaban correlacionados con estratos geográficos.
El análisis de ESTRUCTURA identificó tres subgrupos amplios con estadísticas de diversidad que muestran riqueza y uniformidad similares entre las poblaciones geográficas. Se establecieron dos colecciones núcleo, la colección núcleo tipo 1 (CC-I) y la colección núcleo tipo 2 (CC-X), cada una compuesta por 100 individuos. Estas colecciones fueron significativamente diversas en comparación con el muestreo aleatorio, representaron las principales regiones geográficas y todos los grupos del análisis de la estructura de la población .
El resultado de COLONY confirmó vínculos genealógicos para 241 de 308 muestras, y el 78,0% parecía fiel al tipo. Los análisis de prevalencia de haplotipos asociados a rasgos mostraron que sólo 3943 de los 4033 marcadores se asignaron únicamente al conjunto de F. ×ananassa «Camarosa». La densidad de marcadores fue insuficiente en ciertas regiones para identificar haplotipos asociados a rasgos. En particular, algunas accesiones como «Fletcher» y «L’Amour» poseían múltiples haplotipos de resistencia. Finalmente, se identificaron muestras que contienen haplotipos relacionados con la resistencia FaRCa1, FaRCg1, FaRMp1 y FaRPc2.
En resumen, las colecciones de germoplasma proporcionan una gran cantidad de recursos para que los mejoradores desarrollen y mejoren cultivares. Las colecciones principales creadas en este estudio serán herramientas útiles para identificar nuevos genes y validar la información del ADN para el mejoramiento. Estos hallazgos son valiosos para el mejoramiento y la investigación, ya que brindan una forma más eficiente de utilizar la colección de F. ×ananassa.
Más información: Jason D Zurn et al, Explorando la diversidad y la estructura genética de la Colección Nacional de Fresas Cultivadas de EE. UU., Horticulture Research (2022). DOI: 10.1093/hora/uhac125
