Los científicos han desarrollado un nuevo método para detectar la resistencia a los fungicidas que les permite identificar múltiples mutaciones, tanto conocidas como nuevas, en una sola prueba. Al principio, los investigadores se centraron en proteger los cultivos.
Los fungicidas son los productos agroquímicos más importantes para controlar las enfermedades de los cultivos, de los cuales los fungicidas inhibidores de la desmetilasa o los fungicidas azol se utilizan ampliamente en la protección de plantas.
Los azoles son fungicidas sistémicos de sitio único que se dirigen a la enzima esterol 14α-desmetilasa (CYP51) del citocromo P450, que desempeña un papel importante en la biosíntesis del esterol ergosterol fúngico. Los fungicidas azol alteran la función CYP51 y, por lo tanto, la estabilidad y fluidez de la membrana a través de una combinación de agotamiento de ergosterol y acumulación de intermediarios de esteroles tóxicos.
Los fungicidas azol son muy eficaces y el desarrollo de resistencia tiende a ocurrir más lentamente que con otros fungicidas de sitio único. Sin embargo, se ha informado de un número cada vez mayor de casos de resistencia a los azoles entre patógenos de cultivos económicamente importantes, como Zymoseptoria tritici , que causa el tizón foliar por Septoria en el trigo, Fusarium graminearum y varios otros.
Hay tres mecanismos principales de resistencia a los fungicidas azol: mutaciones en el gen Cyp51 que alteran la unión del fungicida, sobreexpresión de Cyp51 debido a modificaciones del promotor y aumento del flujo de salida de la célula. Estos mecanismos no son mutuamente excluyentes, pudiendo existir varios de ellos en un mismo aislado, aumentando los niveles de resistencia. Otra complicación es la existencia de más de un gen Cyp51 parálogo en ascomicetos filamentosos, incluidos muchos patógenos vegetales. Sin embargo, en ascomicetos con más de un parálogo de Cyp51, las mutaciones asociadas con la resistencia a fungicidas se asocian principalmente con Cyp51A.
Patógenos P. teres f. teres (Ptt) y P. teres f. maculata (Ptm) son los agentes causantes de la mancha neta y la mancha neta de la cebada, respectivamente. Ptt contiene un gen Cyp51B y dos copias de Cyp51A: Cyp51A1 y Cyp51A2, mientras que la mutación de resistencia a fungicidas F489L se encuentra solo en Cyp51A1 10 y hasta la fecha no se han encontrado mutaciones asociadas con resistencia a fungicidas en Cyp51B. A diferencia de Ptt, Ptm contiene solo una copia de Cyp51A, pero las mutaciones del promotor y de la secuencia codificante están asociadas con la resistencia a los fungicidas azol.
Ptt y Ptm también pueden formar híbridos en la naturaleza, y un haplotipo híbrido exhibe altos niveles de resistencia a los fungicidas azólicos, presumiblemente adquiridos mediante recombinación natural entre las dos formas.
Identificar y monitorear la resistencia a los fungicidas tanto en clínicas de agricultura como de medicina humana es fundamental para monitorear las poblaciones resistentes e informar la gestión adecuada.
Por ejemplo, si se detecta resistencia a un compuesto de azol, se puede ajustar el manejo químico para evitar una presión selectiva indebida sobre la población del patógeno, y luego se puede monitorear cuidadosamente a la población para garantizar la efectividad del cambio de tratamiento.
Esto requiere tecnologías de detección molecular rápidas, precisas y accesibles localmente capaces de capturar la compleja red de mutaciones genéticas conocidas y emergentes que causan resistencia. Se han implementado varias tecnologías específicas de alelos, como la amplificación isotérmica en bucle, la PCR digital y la PCR cuantitativa (qPCR), para detectar y monitorear la resistencia a fungicidas en patógenos de plantas.
A pesar de su utilidad, estas tecnologías adolecen de dos limitaciones importantes: la necesidad de análisis separados para cada mutación y la incapacidad de detectar nuevas mutaciones. Hay varias inserciones de promotores diferentes y mutaciones de secuencias codificantes en Ptm que pueden existir solas o en combinación. Ptm y Ptt también están estrechamente relacionados y pueden formar híbridos, lo que complica aún más el diagnóstico de resistencia. Por lo tanto, la detección de resistencia de muestras de campo se convierte en un proceso complejo y que requiere mucho tiempo, e implica una gran variedad de análisis.
Gracias a la inversión conjunta de la Universidad de Curtin y la Corporación Australiana de Investigación y Desarrollo de Granos (GRDC), investigadores del Centro de Enfermedades y Manejo de Cultivos (CCDM) de Australia pudieron detectar de forma rápida y precisa mutaciones de resistencia a fungicidas utilizando un dispositivo portátil de secuenciación de ADN. , MinION de Oxford Nanopore Technologies, Reino Unido, que incluye mutaciones desconocidas que no pueden detectarse con métodos tradicionales.
La investigadora del CCDM y autora principal, la Dra. Katherine Zulak, señaló que los métodos tradicionales para identificar mutaciones dentro de patógenos a menudo requieren mucho trabajo y tiempo y se limitan a detectar sólo mutaciones previamente conocidas.
“La mancha neta de cebada se presenta en dos formas: mancha y red, cada una con su propio conjunto de mutaciones diferentes. Además, el gen diana particular que estudiamos puede tener mutaciones que ocurren en dos regiones diferentes, cada una de las cuales, nuevamente, tiene su propio conjunto de mutaciones. Entonces, cada vez que necesitábamos probar una muestra para detectar resistencia a fungicidas, previamente teníamos que probar cada una de estas posibles mutaciones. Para simplificar este proceso cada vez más complejo, utilizamos Nanopore MinION para secuenciar genes diana de fungicidas y proporcionar un mapa completo de todas las mutaciones posibles, que pueden incluir aquellas que no hemos identificado previamente. Por ejemplo, mientras desarrollábamos este método, descubrimos una variación recién descubierta de una mutación conocida. Este es el tipo de mutación que, de otro modo, podría pasar desapercibida con los métodos tradicionales. El campo de la resistencia a los fungicidas cambia constantemente y esta tecnología simplifica nuestro proceso de identificación de la resistencia a los fungicidas y nos brinda la capacidad de detectar nuevas mutaciones a medida que surgen, lo que nos permite abordar mejor el problema a medida que se desarrolla”, explica el investigador.
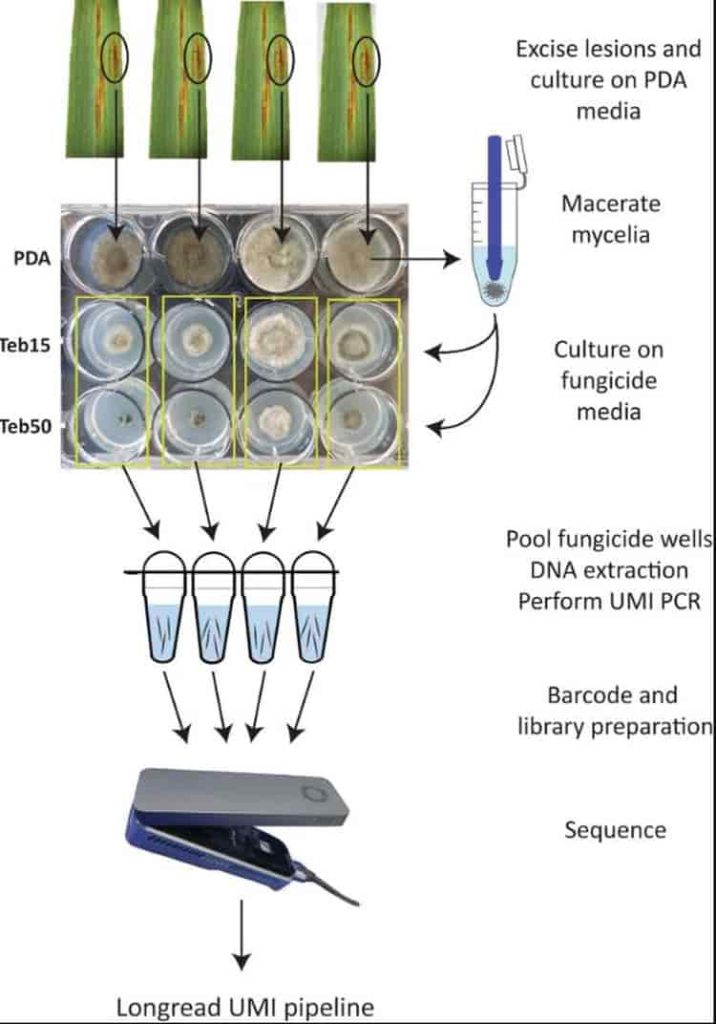
“A medida que secuenciamos regiones del genoma del patógeno, no sólo recopilamos datos para investigaciones en curso, sino que también creamos un recurso para investigaciones futuras. Esta información genética se puede almacenar en bases de datos nacionales, proporcionando orientación a los investigadores en proyectos posteriores y garantizando que los esfuerzos futuros se basen en nuestro conocimiento existente”, afirma el Dr. Zulak.
El profesor Mark Gibberd, director del CCDM, destacó el compromiso del centro de encontrar nuevas herramientas y recursos que puedan acelerar y mejorar la capacidad de la industria para combatir este problema.
«La innovación es el núcleo de nuestro centro de investigación y este proyecto es un gran ejemplo de nuestra capacidad para inspirarnos en una amplia gama de campos para abordar los mayores desafíos de enfermedades de la industria de los cereales», afirmó el profesor Gibberd.
“La inversión en investigación de Australia es sólo una pequeña fracción de la inversión en investigación global. A través de asociaciones internacionales, inversiones conjuntas y colaboraciones CCDM, hemos podido aprovechar la investigación y el desarrollo global para ofrecer innovaciones y soluciones de clase mundial en beneficio de la agricultura australiana”, concluyó.
Fuente: Corporación de Investigación y Desarrollo de Cereales. En la foto: Lina Farfán Cáceres, investigadora asociada de CCDM, y Dra. Katherine Zulak, asociada de investigación, en el laboratorio con MinION de Oxford Nanopore Technologies. Foto: CCDM.
