Investigadores de la Universidad McMaster han desarrollado una nueva forma de prueba rápida para detectar infecciones en animales de granja, en respuesta a la creciente amenaza de brotes peligrosos.
por la Universidad McMaster
El prototipo ha demostrado su eficacia en la detección de una devastadora infección diarreica en cerdos identificada por primera vez en Canadá en 2014, y puede adaptarse para detectar otros patógenos y en otros animales.
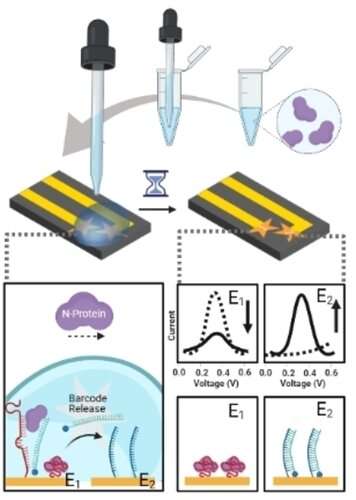
La prueba, creada por la bioquímica Yingfu Li y la ingeniera Leyla Soleymani y sus colegas, utiliza una pequeña muestra de saliva para detectar los marcadores químicos de infección.
Emplea tecnología similar a una forma de prueba que el mismo equipo de investigación creó recientemente para detectar COVID y otras infecciones en humanos. La prueba humana ahora se está moviendo hacia el mercado con fondos públicos de investigación y apoyo corporativo.
Se espera que la prueba en animales, una vez que esté ampliamente disponible, sea una herramienta valiosa para identificar y aislar brotes en granjas, y para limitar la posibilidad de transmisión de infecciones de animales a humanos, que se cree que es el origen de la enfermedad. Pandemia de COVID-19.
Los brotes de enfermedades a menudo requieren la eutanasia de rebaños completos, lo que a veces tiene graves consecuencias económicas y ambientales. Canadá es un productor líder de carne de cerdo, con 14 millones de cerdos en 7600 granjas.
“Realmente existe una clara necesidad de esta tecnología”, dijo Li. “Hay muchas razones por las que todos, incluso las personas que no comen carne de cerdo, deberían preocuparse por la vigilancia de infecciones animales”.
El trabajo ha sido publicado hoy en la influyente revista científica alemana Angewandte Chemie , que lo ha identificado como un “artículo muy importante”, una distinción específica y rara. La investigación fue financiada por el Consejo de Investigación de Ciencias Naturales e Ingeniería de Canadá.
La nueva prueba podría ser un avance significativo en el concepto de “One Health”, la creciente comprensión de la interconexión entre la salud humana, animal y del ecosistema.
La creación de dicha tecnología es parte de la misión del Global Nexus for Pandemics and Biological Threats de McMaster.
Los investigadores diseñaron la prueba basada en aptámeros para que sea portátil, precisa y rápida, lo que permite a los veterinarios y otros cuidadores de animales identificar, aislar y tratar rápidamente a los animales infectados.
La prueba funciona mezclando una pequeña muestra de saliva con un reactivo químico y aplicando la mezcla a un pequeño lector de microchip, que a su vez está conectado a un teléfono inteligente, que muestra los resultados en minutos.
Después de consultar con expertos en el campo, los investigadores optaron por crear su primera prueba en animales para la diarrea epidémica porcina, una amenaza viral grave que puede propagarse rápidamente por granjas enteras.
Uno de los mayores desafíos técnicos en el desarrollo de la prueba durante los últimos cuatro años fue extraer la firma química de la infección de la saliva espesa y a menudo contaminada de los cerdos, utilizando muestras recolectadas por colaboradores veterinarios.
“El desafío aquí fue que las muestras que obtenemos de los hisopos de animales son mucho menos puras que las que obtenemos de los humanos”, dijo Soleymani. “No se le puede decir a un cerdo que se enjuague la boca antes de limpiarlo, así que tuvimos que adaptar nuestro proceso para acomodar estos desafíos”.
Más información: Amanda Victorious et al, Un Aptasensor basado en código de barras de ADN permite la prueba rápida de virus de diarrea epidémica porcina en saliva porcina mediante lectura electroquímica,
Angewandte Chemie International Edition (2022). DOI: 10.1002/anie.202204252
