Los científicos de la Universidad de Nottingham están un paso más cerca de comprender cómo las bacterias, como E. coli y Salmonella enterica, comparten material genético que las hace resistentes a los antibióticos.
por Charlotte Anscombe, Universidad de Nottingham
La resistencia a los antimicrobianos (RAM), la capacidad de los organismos de ser resistentes al tratamiento con antibióticos y otros antimicrobianos, es ahora uno de los problemas más amenazantes en todo el mundo. Las granjas ganaderas, sus entornos circundantes y los productos alimenticios generados a partir de la cría se han destacado como fuentes potenciales de infecciones resistentes para animales y humanos.
En la ganadería , el uso indebido y excesivo de antimicrobianos de amplio espectro administrados para reducir las pérdidas de producción es una importante contribución conocida al gran aumento y propagación de la resistencia a los antimicrobianos.
En este último estudio, los científicos aportan una contribución significativa para demostrar que diferentes especies de bacterias, que coexisten dentro de la misma comunidad microbiana (por ejemplo, dentro del intestino del pollo), son capaces de compartir material genético asociado a la RAM y terminar implementando métodos similares. Mecanismos de resistencia. El descubrimiento tiene implicaciones importantes ya que afecta nuestra comprensión de la RAM y plantea desafíos adicionales para la implementación de soluciones para la vigilancia y el tratamiento/control.
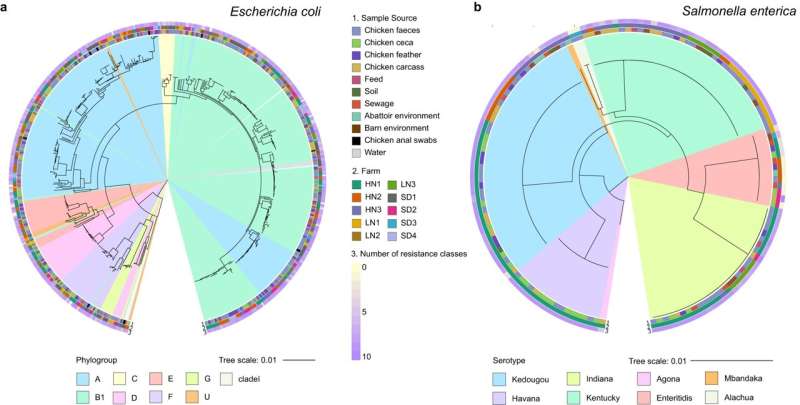
Este estudio, publicado en Nature Communications , analiza dos bacterias importantes que se encuentran en los animales destinados al consumo humano: Escherichia coli y Salmonella enterica, que muestran altos niveles de resistencia a los medicamentos, son comunes en entornos agrícolas, tienen altos niveles de transmisibilidad a los humanos y causan intoxicación alimentaria.
La investigación es una colaboración entre expertos de la Facultad de Medicina y Ciencias Veterinarias de la Universidad, el Centro Nacional de China para la Evaluación de Riesgos de Seguridad Alimentaria, New Hope Liuhe Group Ltd en China y Nimrod Veterinary Products Limited.
La Dra. Tania Dottorini, de la Facultad de Medicina y Ciencias Veterinarias de la Universidad de Nottingham, es la investigadora principal del estudio. Ella dijo: “Estas especies de bacterias pueden compartir material genético tanto dentro como potencialmente entre especies, una forma en la que se propaga la resistencia a los antimicrobianos. Es por eso que comprender hasta qué punto estas bacterias dentro del mismo entorno y, lo que es más importante, en el mismo huésped, pueden coevolucionar y compartir su genoma podría ayudar al desarrollo de tratamientos más eficientes para combatir la RAM”.
El equipo recolectó 661 aislamientos de bacterias de E. coli y Salmonella de pollos y sus entornos en 10 granjas avícolas y cuatro mataderos chinos durante un período de dos años y medio. Llevaron a cabo un análisis a gran escala utilizando métodos de extracción de datos y secuenciación de ADN de microbiología convencional impulsados por el aprendizaje automático.
Este es el primer estudio de este tipo en el que se caracteriza el contenido genómico de dos especies de bacterias a tan gran escala, utilizando muestras recolectadas de los mismos animales, al mismo tiempo y en entornos del mundo real (granjas y mataderos). Los principales hallazgos indican que E. coli y Salmonella enterica que coexisten en el intestino del pollo, en comparación con las que existen de forma aislada, presentan una mayor proporción de material genético relacionado con la resistencia a los antimicrobianos , implementan mecanismos metabólicos y de resistencia más similares, y probablemente sean el resultado de una vía de coevolución más sólida.
El Dr. Dottorini dice: “La aparición y propagación de la resistencia a los antimicrobianos en la ganadería es un fenómeno complejo que surge de una red entrelazada de interacciones que ocurren en múltiples escalas espaciales y temporales y que involucran intercambios entre bacterias, animales y humanos en una multitud de entornos microbianos conectados.
“Invertir en minería de datos y tecnologías de aprendizaje automático capaces de hacer frente a datos heterogéneos a gran escala es crucial para investigar la resistencia a los antimicrobianos, en particular cuando se considera la interacción entre bacterias que cohabitan , especialmente en entornos ecológicos donde se produce una selección de resistencia impulsada por la comunidad.
“En general, este trabajo también ha demostrado que la investigación de especies bacterianas individuales tomadas de forma aislada puede no proporcionar una imagen suficientemente completa de los mecanismos subyacentes a la insurgencia y propagación de la resistencia a los antimicrobianos en la ganadería, lo que podría llevar a una subestimación de la amenaza a la salud humana. “
Más información: Michelle Baker et al, Convergencia de resistencia y respuestas evolutivas en Escherichia coli y Salmonella enterica que cohabitan en granjas de pollos en China, Nature Communications (2024). DOI: 10.1038/s41467-023-44272-1
